Naukowe Walki, czyli jak genetycy wykorzystali współzawodnictwo cząstek?
WCzy w nauce także toczą się batalie? Oczywiście! Rośliny walczące o dostęp do słońca, dzikie zwierzęta wyszarpujące sobie padlinę, pasożyty przystosowane do zabijania gospodarzy, którzy z kolei próbują za wszelką cenę zwalczyć pasożyty… O pierwszeństwo walczą również cząsteczki i atomy – w myśl zasady „kto pierwszy, ten lepszy” łączą się w związki oraz konkurują, na przykład o to, który z nich pierwszy przyłączy się do miejsca aktywnego w enzymie – jak alkohol etylowy i metanol, oba o podobnej budowie. To tylko kilka przykładów naukowych bitew, w których jeden przegrywa by ten drugi mógł wygrać. A czy naukowe batalie może wykorzystać także człowiek po to, by diagnozować i leczyć choroby? Czy takie bitwy toczą się też w … genetyce? Okazuje się, że tak!
Właściwości cząsteczek od dawna zaskakują chemików, fizyków, jak również … biologów. Specjalnie przystosowane związki mogą być wskaźnikami, lekami, bardzo wydajnymi inhibitorami, pełniącymi funkcje hamujące. Połączeniem naukowych batalii i szczególnych cech, jakimi charakteryzują się cząsteczki są dwa znakomicie precyzyjne i bardzo użyteczne testy genetyczne: test fluorescencyjnej hybrydyzacji in situ (FISH), oraz porównawczej hybrydyzacji genomowej do mikromacierzy (aCGH). Pojawiło się dużo trudnych słów i niezrozumiałe nazwy, ale bez obaw – to wszystko jest o wiele prostsze niż mogło by się wydawać!
Głównym problemem, z jakim mierzą się genetycy jest precyzja. Wszystkie geny składają się z tych samych czterech zasad azotowych: adeniny, tyminy, cytozyny i guaniny, ułożonych jak cegiełki w podwójnej helisie DNA. To właśnie od kolejności tych zasad zależy, o czym dany gen decyduje. Aby lokalizować i rozpoznawać geny potrzebna jest znajomość kolejności występujących w nich zasad. Ponieważ Human Genome Project, czyli projekt poznania ludzkiego genomu zakończył się w 2004 roku powodzeniem, mamy już zapisaną kolejność wszystkich zasad w całym genomie ludzkim. Wiemy jakie geny gdzie się znajdują, jaką mają sekwencję par zasad. Znamy ponadto formę, w jakiej „spakowany” jest ludzki genom: to chromosomy, czyli zespiralizowane, mocno ściśnięte, skondensowane DNA. Umiemy je wybarwić, dzięki czemu możemy zaobserwować charakterystyczne prążki na poszczególnych chromosomach. Wydawałoby się, że genom nie jest nam obcy.
Ale co gdy w chromosomach pojawią się aberracje, czyli niezrównoważenia? Co jeśli fragment chromosomu urwie się i będzie go brakować lub podwoi się i będzie go zbyt dużo? Albo dwa fragmenty chromosomów zamienią się miejscami? Wtedy można spróbować zaobserwować zaburzony układ chromosomowych prążków. Wprawne oko doświadczonego genetyka dostrzeże zmiany w ich ilości lub kolejności i będzie w stanie ocenić stratę, dodatek lub miejsca zamiany. Ale co jeśli zmiany będą mniejsze niż wielkości, które można obserwować na prążkach? Co jeśli zamienią się miejscem właśnie fragmenty pomiędzy prążkami lub same końcówki chromosomów?
Cóż, trzeba przyznać, że ten problem spędzał genetykom sen z powiek przez dłuższy czas. Jak wykryć translokacje, delecje i duplikacje o wielkościach mniejszych niż te, które można oglądać pod mikroskopem, czyli zmiany submikroskopowe? Jak zajrzeć w geny na tak małym poziomie? Walka
z tajemniczością chromosomów trwała i trwała, aż w końcu… właśnie walka cząsteczek rozwiązała ten problem.
Wyobraźmy sobie wielkie pole. Na tym polu, jedna przy drugiej, zostały ustawione wysokie wieże zbudowane w całości z czterech rodzajów klocków. Klocki powtarzają się, występując w każdej wieży w odmiennej kolejności, przy czym każdy klocek ma z jednej strony specjalnie przygotowane połączenia: dwa z czterech klocków mają trzy druciki, pozostałe dwa mają po dwa druciki. To genom ludzki, pocięty na mniejsze kawałki. Podwójna helisa została rozerwana, teraz pozostały już pojedyncze, krótsze nitki. Klocki w wieży to zasady – adenina i tymina mogą tworzyć po dwa wiązania, cytozyna i guanina mogą tworzyć po trzy. Teraz wyobraźmy sobie, że na nasze pole spada deszcz nowych wież; różnią się one od wież z pola – są kolorowe: połowa wież jest niebieska, a druga połowa czerwona. Klocki w tych wieżach nie różnią się jednak od klocków wież z pola: także tworzą po dwa i trzy połączenia. Co zatem zrobią wieże spadające na pole z deszczem? Będą, a jakże, walczyć – o to, które pierwsze przyłączą się do wież z pola: czerwone czy niebieskie.
Ta wieżowa metafora to tak naprawdę opis testu aCGH, czyli porównawczej hybrydyzacji genomowej do mikromacierzy. Wieże z pola, bezbarwne, to oligonukleotydy. Za tą trudną nazwą kryje się określenie DNA człowieka pokrojonego na mniejsze kawałki i umieszczonego na płytce macierzy przez specjalne urządzenie. Kolorowe wieże to także pokrojone na kawałki DNA, ale innego pochodzenia. Czerwone wieże to fragmenty DNA pobranego od pacjenta. Genetyk nie wie, co dolega tej osobie – nie wie, czy ma ona jakieś delecje lub duplikacje. Natomiast niebieskie wieże
to fragmenty tak zwanego DNA kontrolnego, inaczej referencyjnego. Jest ono poprawne, ma znaną, niezaburzoną kolejność zasad i stanowi dla badacza odnośnik względem DNA od pacjenta. Gdy jedne i drugie oligonukleotydy zostają umieszczone wraz z roztworem na płytce, konkurują one o to, które przyłączą sie do fragmentów już obecnych na macierzy. Gdyby okazało się, że materiału od pacjenta i materiału kontrolnego jest tyle samo, kolor widoczny na komputerowym obrazie ze skanera byłby połączeniem kolorów barwników zastosowanych na jednych i drugich nitkach. To jasne, ale w jaki sposób taka kombinacja pomoże naukowcowi odnaleźć submikroskopowe zmiany? To całkiem proste. Wyobraźmy sobie, że pacjent ma delecję – brakuje mu fragmentu chromosomu. Wtedy, po pocięciu chromosomu na mniejsze kawałki okaże się, że części brakuje. Ale w DNA kontrolnym te oligonukleotydy oczywiście występują, ponieważ jest ono poprawne. Jaki zatem będzie efekt? Fragmenty DNA kontrolnego będą przyłączać się do odpowiadających im nici na macierzy i nie będą mieć przeciwwagi w postaci fragmentów DNA od pacjenta. Dlatego kolor DNA kontrolnego w danym punkcie macierzy będzie dominować. Dzięki temu superczuły (i jednocześnie bardzo drogi) skaner będzie mógł z tej nierówności kolorystycznej odczytać, w którym miejscu na chromosomie pacjenta brakuje części genów. Z drugiej strony, jeśli u pacjenta pojawi się duplikacja, w niekorzystnej sytuacji znajdzie się DNA kontrolne. Oligonukleotydów od pacjenta, ze względu na duplikację, czyli powiększenie ilości danych genów, będzie po prostu więcej i dlatego będą one miały większe prawdopodobieństwo przyłączenia sie do nici z macierzy. Tak samo jak wcześniej, spowoduje to nierównomierne rozłożenie koloru w tym punkcie macierzy, tylko że tym razem przebijającym kolorem będzie kolor DNA pacjenta. Tę prawidłowość także może odczytać i zanalizować skaner, dając badaczowi możliwość zorientowania się, gdzie w DNA występuje błąd.

Metoda hybrydyzacji porównawczej jest znakomita jeśli chodzi o rozpoznawanie strat i nabytków genetycznych. Co jednak jeśli chcemy się dowiedzieć jak taka zmiana się pojawiła lub jeśli podejrzewamy translokację zrównoważoną? Test aCGH daje możliwość oceny jedynie ilościowej, ogranicza natomiast możliwość lokalizacji pochodzenia utraconych fragmentów lub przestrzennego zobrazowania ułożenia genów na chromosomach.
Co zatem zrobić jeśli wykryjemy u pacjenta małą delecję i duplikację i chcemy dowiedzieć się, co stało się z tymi zaburzonymi fragmentami chromosomów, gdzie one się znajdują? Wtedy możemy zastosować inny test – test FISH, czyli fluorescencyjnej hybrydyzacji in situ.
Gdy wykonamy test aCGH, poznajemy dokładną lokalizację genów, których brakuje lub które zostały podwojone. Tę wiedzę możemy wykorzystać do wytworzenia specjalnych sond – znaczników, mających za zadanie przyłączenie się do określonych fragmentów chromosomów – właśnie tych utraconych lub podwojonych. Sondy takie uzyskujemy z klonów bakteryjnych. Po zakończeniu Human Genome Project stworzono specjalne bakteryjne biblioteki DNA. Bakterie posiadają jeden chromosom bakteryjny oraz dodatkową, małą cząsteczkę kolistego DNA, zwaną plazmidem.
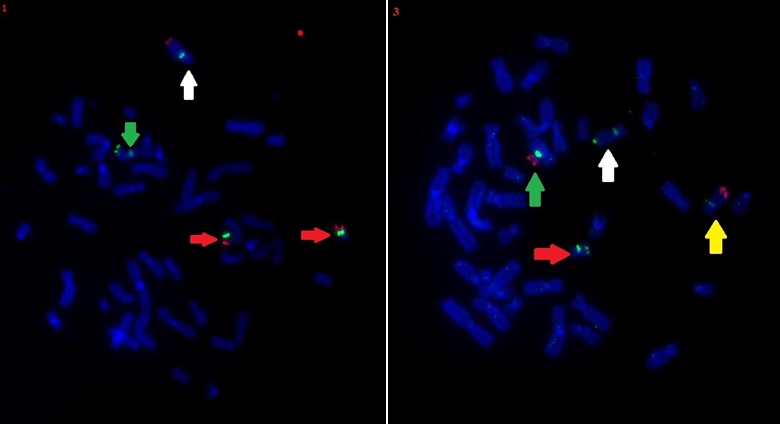
Do plazmidu łatwo jest „włożyć” obce DNA, na przykład fragment DNA pobranego od człowieka. Dlatego ludzkie DNA pocięto na mniejsze części i umieszczono w plazmidach bakterii, aby w razie potrzeby móc łatwo się posługiwać tymi mniejszymi kawałkami i użyć ich, gdy okażą się potrzebne. Tak też dzieje się w przypadku testu FISH: z bakterii badacz izoluje potrzebny mu fragment DNA ludzkiego – ten, którego zaburzoną ilość wykazał test aCGH. Może łatwo go odnaleźć, gdyż plazmidy są cyfrowo skatalogowane i opisane. Następnie, za pomocą specjalnych barwników, nadaje kolor utworzonym przez siebie sondom. Jednocześnie z komórek pobranych od pacjenta przygotowuje preparaty na płytce – są to zatrzymane w trakcie podziału chromosomy, częściowo rozwinięte – podwójna helisa DNA jest rozdzielona, nici pozostają osobno i sondy mogą się do nich przyłączyć. Następnie, w specjalnych warunkach, badacz nakłada na płytkę przygotowane sondy i pozostawia je do hybrydyzacji, czyli do łączenia. Jaki efekt uzyskuje? Sondy przygotowane z bakterii pasują do zaburzonych fragmenty chromosomów – to znaczy, że zawarty w nich fragment DNA z biblioteki bakteryjnej jest komplementarny, czyli może się przyłączyć tylko do tego fragmentu DNA pacjenta, którego zaburzoną ilość wykrył test aCGH. Zatem, jeżeli sonda jest wycelowana w utracony fragment chromosomu, na przykład, 10., pod mikroskopem nie otrzymamy żadnego obrazu – na chromosomie 10. fragment ten nie będzie obecny. W ten sposób można łatwo zweryfikować poprawność wykonanego już wcześniej testu aCGH.
Jednak test FISH daje także inne możliwości. Gdy u pacjenta pojawi się duplikacja, często ten powielony fragment zmienia swoje położenie i przyczepia się do innego chromosomu. Ponieważ kolejność genów się w nim oczywiście nie zmienia, sonda nadal „pasuje” do tego fragmentu, zatem przyłącza się do niego i daje fluorescencję, czyli efekt barwny, podczas obserwacji pod mikroskopem. Możemy zatem zaobserwować, gdzie powędrował ten powielony kawałek DNA i w jakim miejscu się teraz znajduje. Co więcej – gdy bada się pacjentów pokoleniami, na przykład oboje rodziców oraz ich dziecko, można dzięki testowi FISH wykryć pochodzenie zmian genetycznych. Na przykład dziecko może odziedziczyć niezrównoważenie genetyczne po jednym z rodziców, który, choć był zdrowy, miał w swoim genomie translokację wzajemną zrównoważoną. U ojca lub matki nie będzie zmienionej dawki genów – dlatego nie będzie też żadnych zaburzeń. Jednak gdy powstaje zygota, a z niej zarodek, z których później ukształtuje się organizm dziecka, dziedziczy ono tylko po jednym chromosomie z każdej pary od każdego z rodziców. Od rodzica z translokacją może zatem odziedziczyć albo dwa zdrowe chromosomy, albo te dwa z translokacją – i wtedy także będzie zdrowe. Jednak, jak wiadomo, dobór chromosomów które trafiają do jądra komórkowego zygoty jest losowy: dziecko może więc także otrzymać jeden chromosom zdrowy i jeden chory. Wtedy będzie ono wykazywać objawy chorobowe. Badacz natomiast, wykonując test aCGH dziecka i rodziców wykryje niewłaściwą ilość genów tylko w genomie dziecka. Jeśli jednak wykona także test FISH, będzie mógł zaobserwować pod mikroskopem niewłaściwie sygnały pochodzące z zaburzonych chromosomów zarówno u rodziców, jak i u dziecka.
Przykład odrobinę rozjaśni tę prawidłowość. Wyobraźmy sobie, że pacjent, dziecko, ma delecję na chromosomie 10. i duplikację na chromosomie 20. Oznacza to, że brakuje mu fragmentu chromosomu 10., ma natomiast zbędny, dodatkowy kawałek chromosomu 20. Gdy wykonamy test FISH tego dziecka, widzimy, że ten dodatkowy kawałek chromosomu 20. jest przyłączony w miejscu brakującego fragmentu chromosomu 10. Potem wykonujemy test FISH matki i otrzymujemy wynik – matka ma translokację zrównoważoną. Końcówka jednego z jej chromosomów o numerze 10. zamieniła się z końcówką jednego z jej chromosomów 20. Ponieważ ojciec pacjenta jest zdrowy, dziecko otrzymuje od niego dwa zdrowe chromosomy. Ale od matki może otrzymać różne kombinacje i losowo otrzymuje akurat zmieniony chromosom 10. z końcówką chromosomu 20. i zdrowy chromosom 20. W ten sposób powstaje jego zaburzenie – ale wykrycie przyczyny tego zjawiska było możliwe dopiero dzięki testowi FISH.

Testy FISH i aCGH wykorzystują naturalną skłonność cząsteczek do rywalizacji o to, które z nich pierwsze utworzą wiązania. W przypadku tego pierwszego testu cząstki walczą o przyłączenie się do DNA zawartego na matrycy, w tym drugim teście próbują połączyć się z odpowiadającymi sobie fragmentami na chromosomach. Dzięki tym właściwościom badacze mają szansę diagnozować pacjentów, zaglądać w ich genom i odkrywać przyczyny nieprawidłowości. Przemyślność mechanizmów przyrody może zatem być wykorzystana także przez ludzi do uzyskania wymiernych korzyści, dzięki którym dowiadujemy się więcej o nas samych. Zatem walka o pierwszeństwo wcale nie musi być taka negatywna, jak mogłoby się nam wydawać, a najlepiej wiedzą o tym genetycy, walczący na co dzień z tajemnicami ukrytymi w kolejności tych czterech niepozornych azotowych klocków, na których zbudowany jest cały żywy świat…
Praca Konkursowa: Dalia Gala